Chủ đề thịnh hành
#
Bonk Eco continues to show strength amid $USELESS rally
#
Pump.fun to raise $1B token sale, traders speculating on airdrop
#
Boop.Fun leading the way with a new launchpad on Solana.
Cần gì để một máy tính học các quy tắc ghép cặp RNA?
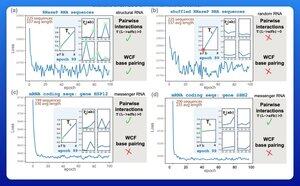
Mọi người đang đào tạo các mô hình ngôn ngữ lớn để dự đoán cấu trúc RNA. Một số mô hình này có hàng trăm triệu tham số.
Một kết quả sớm thú vị là những mô hình này học các quy tắc ghép cặp Watson-Crick-Franklin trực tiếp từ dữ liệu.
Một nhóm nghiên cứu tại Harvard đã quyết định xem mô hình nhỏ nhất có thể đạt được kết quả này là gì.
Họ đã đào tạo một mô hình xác suất nhỏ với chỉ 21 tham số bằng cách sử dụng phương pháp giảm dần độ dốc.
Với chỉ 50 chuỗi RNA—không có cấu trúc tương ứng—các quy tắc ghép cặp sẽ xuất hiện sau chỉ vài vòng đào tạo.
Vì vậy, câu trả lời cho câu hỏi ban đầu của họ là "ít hơn nhiều so với bạn nghĩ" để học loại mô hình này.
Tôi không nghĩ điều này có nghĩa là các nỗ lực đào tạo quy mô lớn là ngu ngốc hoặc sai lầm. Nhưng kết quả này gợi ý rằng còn rất nhiều hiệu quả và hiệu suất có thể được khai thác từ sự đổi mới trong kiến trúc.
Có rất nhiều cấu trúc cơ bản trong ngôn ngữ của sinh học.
3,35K
Hàng đầu
Thứ hạng
Yêu thích













