Popularne tematy
#
Bonk Eco continues to show strength amid $USELESS rally
#
Pump.fun to raise $1B token sale, traders speculating on airdrop
#
Boop.Fun leading the way with a new launchpad on Solana.
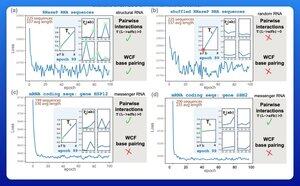
Co jest potrzebne, aby komputer nauczył się zasad parowania zasad RNA?
Ludzie trenują duże modele językowe do przewidywania struktury RNA. Niektóre z tych modeli mają setki milionów parametrów.
Jednym z ekscytujących wczesnych wyników było to, że te modele uczą się zasad parowania zasad Watsona-Cricka-Franklina bezpośrednio z danych.
Grupa badawcza z Harvardu postanowiła sprawdzić, jaki jest najmniejszy możliwy model, który mógłby osiągnąć ten wynik.
Wytrenowali mały model probabilistyczny zaledwie z 21 parametrami, używając spadku gradientu.
Przy tak niewielkiej liczbie jak 50 sekwencji RNA—bez odpowiadających struktur—zasady parowania pojawiały się po zaledwie kilku epokach treningowych.
Zatem odpowiedzią na ich pierwotne pytanie było to, że potrzeba "dużo mniej, niż mogłoby się wydawać", aby nauczyć się tego typu modelu.
Nie sądzę, aby to oznaczało, że duże wysiłki w zakresie treningu są koniecznie głupie lub błędne. Ale ten wynik sugeruje, że istnieje wiele efektywności i wydajności, które można jeszcze uzyskać dzięki innowacjom architektonicznym.
Jest wiele ukrytej struktury w języku biologii.
3,25K
Najlepsze
Ranking
Ulubione













