Tópicos em alta
#
Bonk Eco continues to show strength amid $USELESS rally
#
Pump.fun to raise $1B token sale, traders speculating on airdrop
#
Boop.Fun leading the way with a new launchpad on Solana.
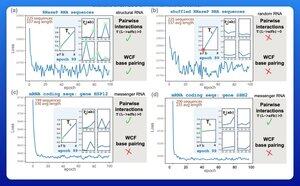
O que é preciso para um computador aprender as regras do emparelhamento de bases de RNA?
As pessoas estão treinando grandes modelos de linguagem para previsão da estrutura do RNA. Alguns desses modelos têm centenas de milhões de parâmetros.
Um resultado inicial empolgante foi que esses modelos aprendem as regras do emparelhamento de bases Watson-Crick-Franklin diretamente dos dados.
Um grupo de pesquisa em Harvard decidiu ver qual era o menor modelo possível que poderia alcançar esse resultado.
Eles treinaram um pequeno modelo probabilístico com apenas 21 parâmetros usando gradiente descendente.
Com apenas 50 sequências de RNA - sem estruturas correspondentes - as regras do emparelhamento de bases apareceriam após apenas algumas épocas de treinamento.
Portanto, a resposta à pergunta original foi que é preciso "muito menos do que você imagina" para aprender esse tipo de modelo.
Não acho que isso signifique que os esforços de treinamento em grande escala sejam necessariamente estúpidos ou equivocados. Mas esse resultado sugere que há muita eficiência e desempenho que ainda podem ser obtidos com a inovação arquitetônica.
Há muita estrutura subjacente à linguagem da biologia.
3,21K
Melhores
Classificação
Favoritos













