トレンドトピック
#
Bonk Eco continues to show strength amid $USELESS rally
#
Pump.fun to raise $1B token sale, traders speculating on airdrop
#
Boop.Fun leading the way with a new launchpad on Solana.
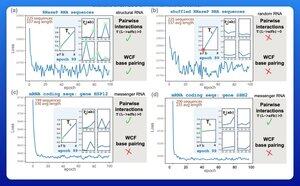
コンピューターがRNA塩基対のルールを学習するには何が必要ですか?
人々は、RNA構造予測のために大規模な言語モデルをトレーニングしています。これらのモデルの中には、数億のパラメータを持つものもあります。
興味深い初期の結果は、これらのモデルがデータから直接ワトソン-クリック-フランクリン塩基対のルールを学習することです。
ハーバード大学の研究グループは、この結果を達成できる可能な限り最小のモデルが何であるかを確認することにしました。
彼らは、勾配降下法を使用して、わずか 21 個のパラメーターを持つ小さな確率モデルをトレーニングしました。
対応する構造がない 50 個の RNA 配列では、塩基対のルールは、わずか数回のトレーニング エポックで明らかになります。
したがって、彼らの最初の質問に対する答えは、このタイプのモデルを学ぶのに「思っているよりもはるかに少ない」というものでした。
これは、大規模なトレーニングの取り組みが必ずしも愚かで見当違いであるという意味ではないと思います。しかし、この結果は、アーキテクチャの革新からまだ多くの効率とパフォーマンスが得られることを示唆しています。
生物学の言語には多くの根底構造があります。
3.22K
トップ
ランキング
お気に入り













