Актуальные темы
#
Bonk Eco continues to show strength amid $USELESS rally
#
Pump.fun to raise $1B token sale, traders speculating on airdrop
#
Boop.Fun leading the way with a new launchpad on Solana.
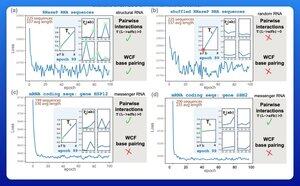
Что нужно, чтобы компьютер научился правилам паросочетания оснований РНК?
Люди обучают большие языковые модели для предсказания структуры РНК. Некоторые из этих моделей имеют сотни миллионов параметров.
Одним из захватывающих ранних результатов стало то, что эти модели учат правила паросочетания оснований Уотсона-Крика-Франклина непосредственно из данных.
Исследовательская группа в Гарварде решила выяснить, какая самая маленькая модель может достичь этого результата.
Они обучили крошечную вероятностную модель всего с 21 параметром, используя градиентный спуск.
С помощью всего 50 последовательностей РНК — без соответствующих структур — правила паросочетания проявлялись уже после всего нескольких эпох обучения.
Таким образом, ответ на их первоначальный вопрос заключался в том, что для обучения этой модели требуется "гораздо меньше, чем вы могли бы подумать".
Я не думаю, что это означает, что усилия по обучению на большом масштабе обязательно глупы или ошибочны. Но этот результат предполагает, что существует много эффективности и производительности, которые все еще можно извлечь из инноваций в архитектуре.
В языке биологии есть много скрытой структуры.
3,39K
Топ
Рейтинг
Избранное













